DArTag は、任意のSNP/Indelマーカーを標的として、数百~数千マーカーのサイズのジェノタイピングを行うことができるDArT社(Diversity Arrays Technology P/L社)独自の手法です。マーカー探索(WGS、RAD-seq、GRAS-Di、等)で取得した全ゲノムSNPの中から、選抜したご希望のSNPについて、ジェノタイピングを行うことができます。
※解析はDiversity Arrays Technology P/L社で実施します。

特長
・QC後1~1.5か月で、ジェノタイピング結果を納品。
・構築したパネルは、新たなマーカーを追加したり、不要なマーカーを削除できる。
・マイクロハプロタイプを検出できる。
・GBSとは異なり、欠損データ率が低く、新しいデータを既存のデータに簡単に追加できる。
・DArTagの取得リード数量は、GBS(数Mreads)のように多くなく、数十kreads以下であるため、下流のバイオインフォマティクス処理(連鎖地図作成、QTL解析、Marker Assisted Selection、ゲノム予測)の計算が軽くなる。
解析の流れ

パネル構築
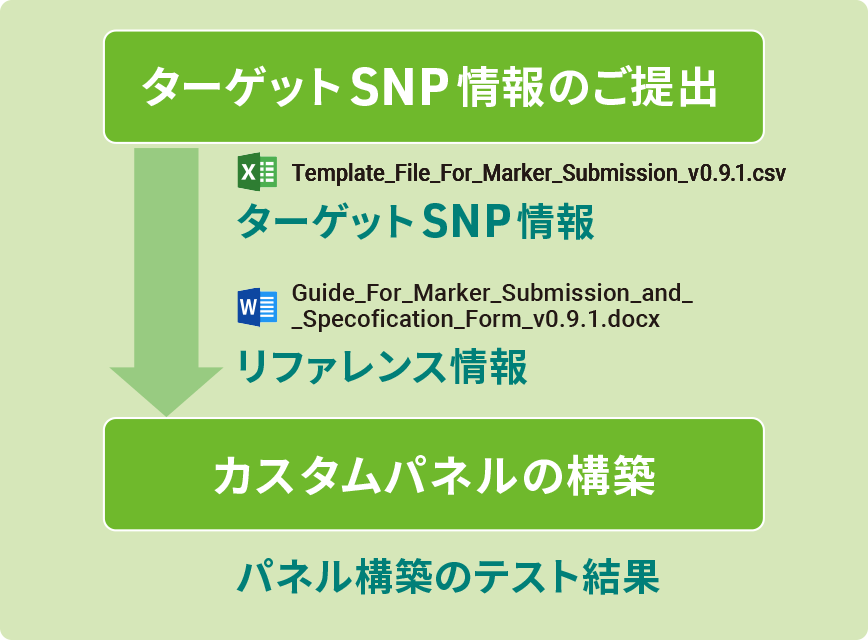
マーカー設計の段階では、ターゲットSNP情報に含まれるマーカーが、DArT社独自のマーカー設計および QC パイプラインを通じて処理されます。
パネル構築に際しては、以下の情報をご提供頂きます。
・ターゲットSNP情報
・リファレンス情報
既存パネルの利用
構築済みのパネルが用意されている生物種については、既存のパネルをご利用頂く事も可能です。詳しくはご相談ください。

ジェノタイピング
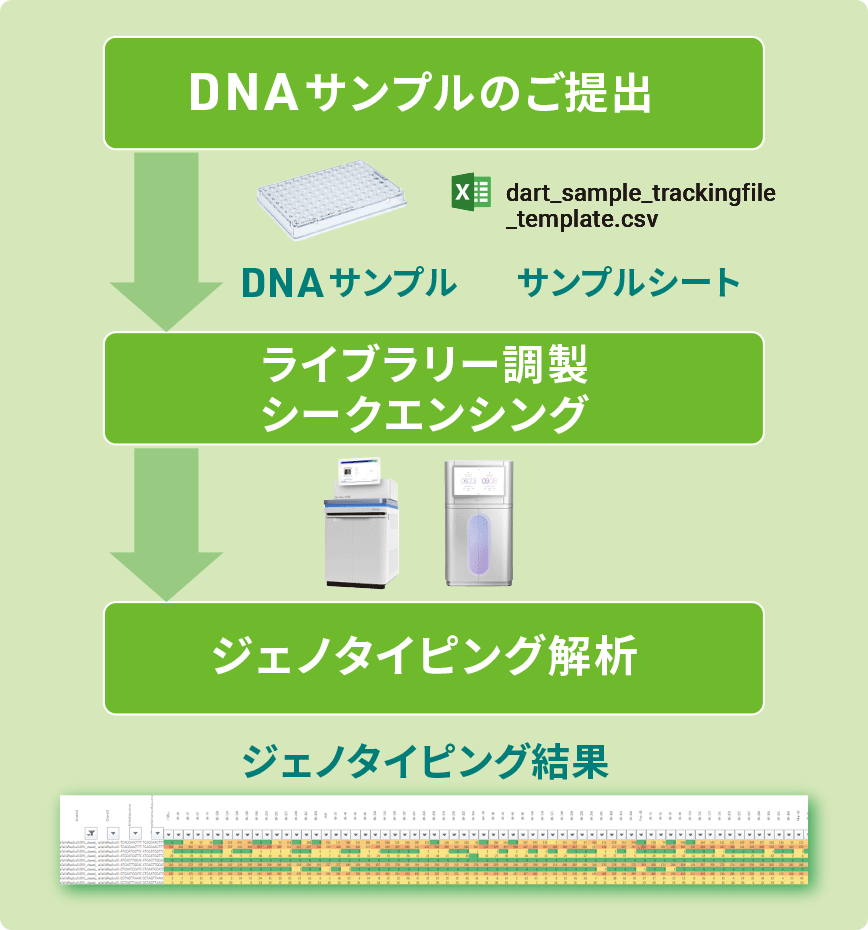
DArTag は、SNP/Indelをキャプチャした後、ターゲット領域を増幅してライブラリーを作製し、Illuminaシーケンスとジェノタイピング解析を⾏います。
既存⼿法より⾼効率な⽅法で、パネル当たり最⼤4,000マーカーのジェノタイピングを⾏うことができます。
1マーカーあたりのDepthは通常100〜300Xですが、ゲノムサイズ、集団の遺伝的多様性、倍数性、等によって異なります。
データ形式
DArTagでは、2 種類のデータが生成されます。
- 【優性マーカー】
-
PCR 増幅の有り無しで表現されるスコアで、SilicoDArTと呼びます。DArTagで取得したシーケンスデータからin silicoで抽出されます。

- 【共優性マーカー】
-
SNP抽出情報です。リファレンスホモ:0、ヘテロ:1、変異ホモ:2として表記されます。
解析事例
アルファルファ(Medicago sativa L.)の例
Zhao D, et al. (2023). Genetic Resources 4:55–63.
A public mid-density genotyping platform for alfalfa (Medicago sativa L.).
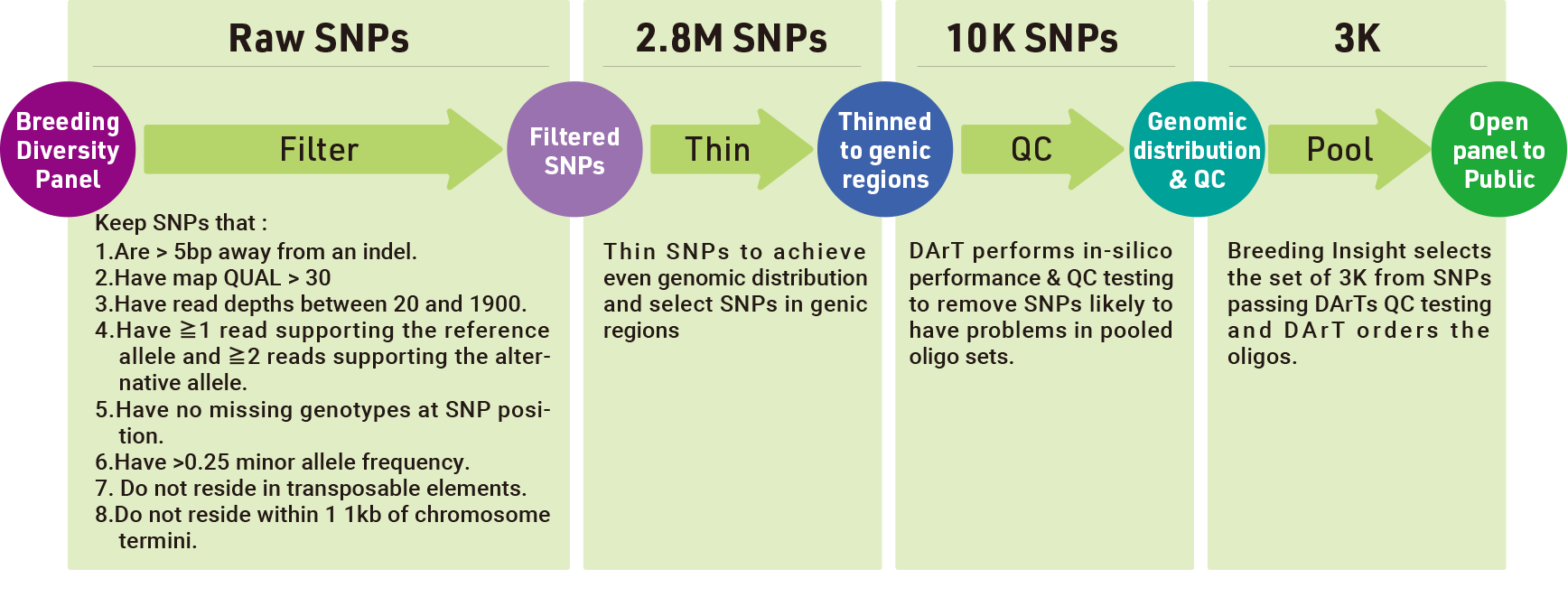
40個体の全ゲノムシークエンスを行ってSNPを取得した後、フィルタリングで2.8M SNPに絞りました。2.8M SNPをさらに間引いて、ゲノム分布の均一化と遺伝子領域におけるSNPの最大化を図り、10K SNPセットを作成しました。最終的に、10K SNPの中からDArT社のQCに合格した3K SNPを選択して、オリゴDNAを合成しました。
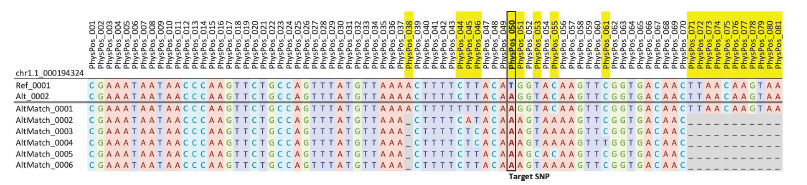
Chr1.1_000194324遺伝子座におけるDArTagシーケンスリード。
DArTagアッセイは、Target SNPを検出して、Reference AlleleとAlternative Alleleを区別できるように設計されています。各配列は、パネルで検出されたマイクロハプロタイプです。追加の変異位置によって、個々のマイクロハプロタイプが区別されます。
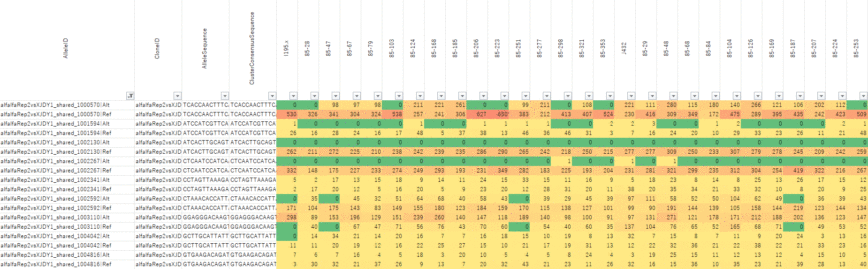
DArTagによるジェノタイピング結果。
行はSNPマーカー、列はサンプルを表し、各列のリード数がPCR増幅の有り無しを表します。
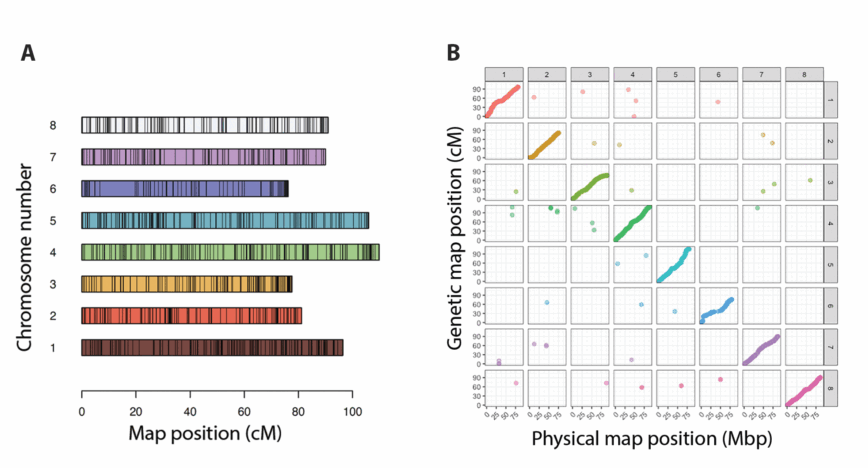
DArTagジェノタイピング結果を基に作成した連鎖地図。
最初に、F1集団とBC1集団で個別に遺伝子地図を構築した後、遺伝位置を基にマージして得られた連鎖地図です。DArTagで取得したマーカーは、アルファルファの染色体数と同じく、8つの連鎖群に分かれました。
物理位置と遺伝位置の相関の高い遺伝地図を構築することができ、マッピングされたすべてのマーカーのうち、F1マップで異なる染色体に割り当てられたのはわずか2.55%、BC1マップで異なる染色体に割り当てられたのは0.97%でした。
価格・納期
サンプル要件
DNAサンプル
- 液量40ul以上推奨(蛍光測定で濃度50〜100ng/uL)
- DNAの分解が少ないこと
- 不純物/蛍光⾊素/RNA/タンパク質の混⼊が無いこと
Positive Control
- 液量500ul以上必須(蛍光測定で濃度〜25ng/uL)
- DNAの分解が少ないこと
- 不純物/蛍光⾊素/RNA/タンパク質の混⼊が無いこと
- EBバッファー(10mM Tris-Cl, pH8.5)に溶解


