特長
PacBioシーケンスはPacific Biosciences社で開発されたシーケンス手法で、1分子リアルタイム(SMRT:Single Molecule Real Time)シーケンシング技術を用いることで、高品質のロングリードシーケンスデータを得ることが出来ます。

解析用途
- 新規ゲノム配列決定
- SNV / Indel フェージング解析
- トランスクリプトーム解析
- 構造変異解析
- メチル化解析
- 融合遺伝子解析
PacBioシーケンスの原理
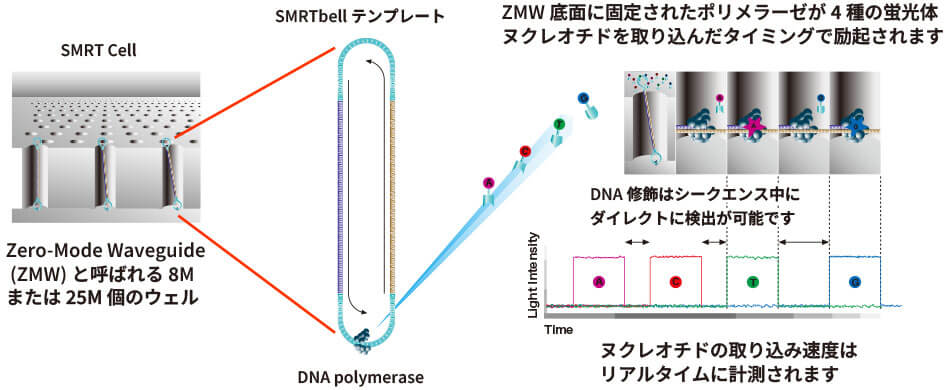
Pacific Biosciences社のシーケンサーでは、DNAポリメラーゼの伸長反応において取り込まれた4種の蛍光体dNTPから放出される蛍光体ピロリン酸をリアルタイムに計測することにより、鋳型DNAの塩基配列を決定します。
鋳型となる二本鎖DNAは、ヘアピンループ型のアダプターを両末端に結合することで、SMRTbellテンプレートと呼ばれる一分子二本鎖DNAライブラリーに変換されます。
ライブラリーは、SMRT Cell上に数百万個以上存在するZMW(zero-mode waveguides)と呼ばれるウェルにロードされ、ウェルの底面に固定されたDNAポリメラーゼによりシーケンシングされます。
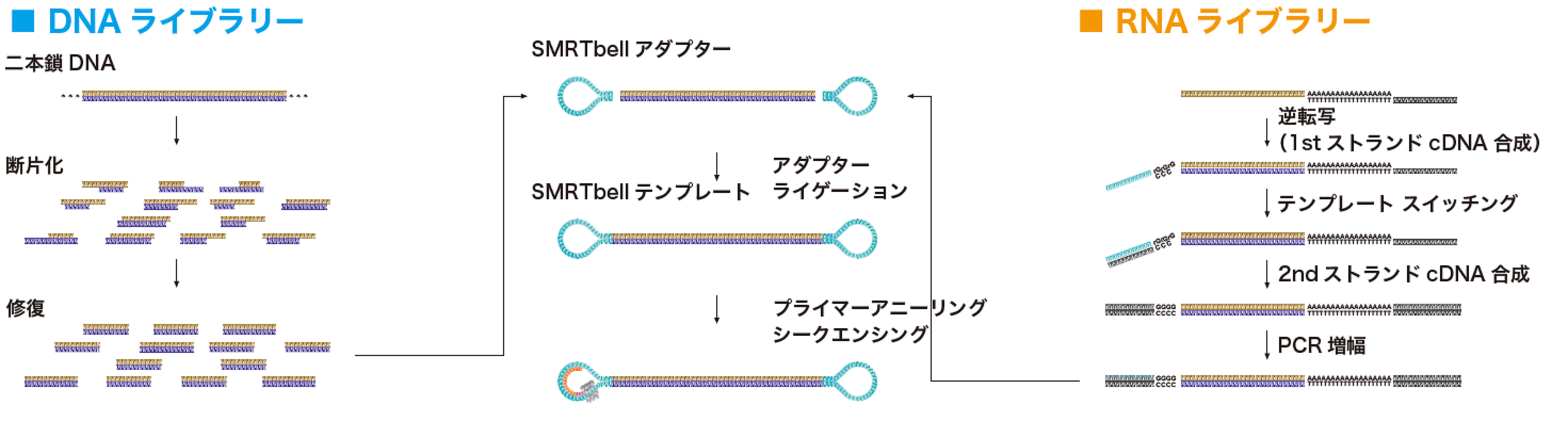
ライブラリー調製
DNA またはRNA サンプルから以下の手順でライブラリー(SMRTbell テンプレート)を調製します。
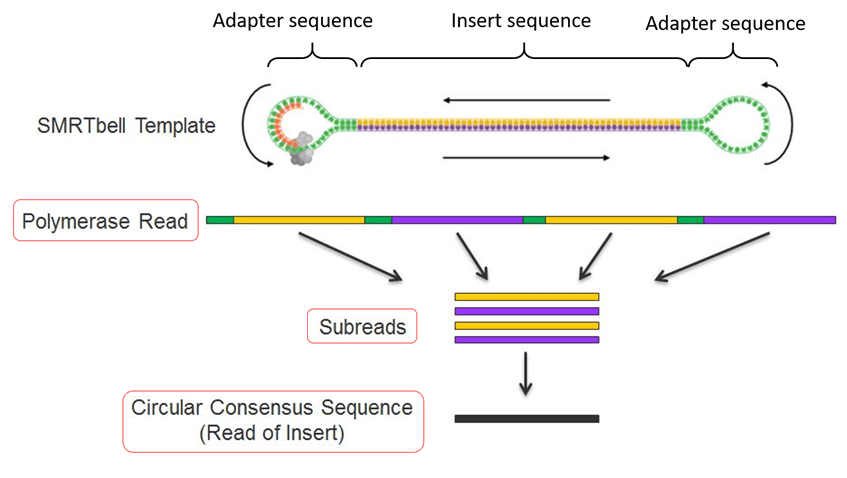
PacBioシーケンスデータ
PacBioシーケンシングによって得られるリード配列には複数の段階があり、それぞれ異なる名前で呼ばれます。
ポリメラーゼリード(Polymerase Read)
シーケンス反応により最初に出力されるアダプターを含んだリード配列
サブリード(Subreads)
ポリメラーゼリードからアダプター配列を除去し、分割されたインサートのみのリード配列
CCSリード(Circular Consensus Sequence)
同一ポリメラーゼリード由来のサブリードのコンセンサス配列
HiFiリード(High-Fidelity Long Read)
CCSリードのうち、Q20(99%)を越える精度を持つリード配列

解析の流れ
以下のサンプルをお受入れ可能です。
- 抽出済みDNA
- 抽出済みRNA
- PCR増幅産物
- 細胞 / 血液 / 組織検体
- 調製済みSMRTbell テンプレート
サンプル要件
- DNA
必要サンプル量:7.5ug(推奨15ug)
濃度 200ng/ul以上、液量50ul以上
分解が少ないこと - RNA
必要サンプル量:1ug(推奨3ug)
濃度 50ng/ul以上、液量20ul以上
RIN値 7以上、rRNA ratio 1.5以上
ご送付いただいたサンプルのクオリティを確認いたします。
各種アプリケーション用のライブラリーを調製いたします。
PacBio Sequel IIe または Revioシステムでのシーケンス
オプションで各種データ解析を承ります。

