DNAナノボール技術による、
新しいタイプのショートリードシーケンス
新しいタイプのショートリードシーケンサーであるMGI Tech社のDNBSEQ-G400システムおよびDNBSEQ-T7システムを用いたシーケンス受託解析サービスを提供いたします。
DNBSEQは環状化された一本鎖DNAをテンプレートとし、ローリングサークル増幅(RCA)によってライブラリーを増幅し、塩基配列を解読します。PCR-Freeライブラリーを選択すれば、すべての解析工程において、PCR反応を一切行わず解析することができるため、従来型のショートリードシーケンサーと比べて、以下の点で優れています。

- PCR重複リードの抑制
- ライブラリー調製時や増幅時のPCRエラーの蓄積による配列読み取りエラーの軽減
- PCR増幅時のGCバイアスの減少
DNBSEQの原理
(1)一本鎖DNAの環状化
末端にアダプター配列をライゲーションさせた二本鎖DNAを加熱して変性させ、一本鎖DNAにします。
5’ および 3’の両末端のアダプター配列に相補的な配列を持つオリゴヌクレオチドをハイブリダイズさせ、ニックを修復することにより、環状一本鎖DNA(ssCirDNA)を調製します。
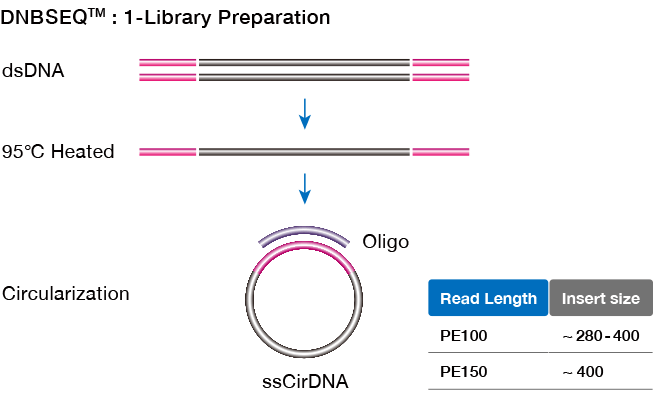
(2)DNAナノボール
環状化した一本鎖DNAをテンプレートととしてローリングサークル増幅することにより、約100〜1000コピーの配列がつながったDNAナノボールを形成します。
増幅されるDNAは毎回同じ環状化一本鎖DNAをテンプレートとするため、約100〜1000コピーの全てに同じ位置でエラーが入ることが抑止可能となります。
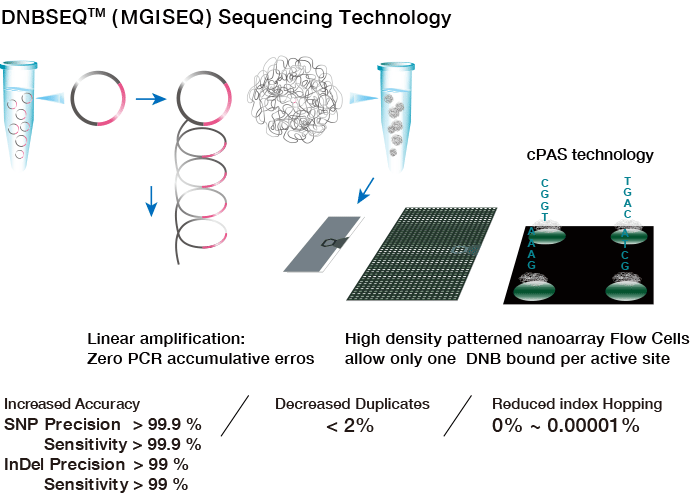
(3)シーケンシング
DNAナノボールは、酸性条件では負の電荷を帯びます。シリコンチップの表面にパターン化された結合サイトは正の電荷を帯びており、この負と正の相互作用により、DNAナノボールがスライド表面にロードされます。
シーケンシングはライブラリーキットやアプリケーションにより、シングルエンド50bp、400bp、ペアエンド100bp、150bp、200bpに対応しています(2021年3月時点)。
(4)データ形式
シーケンスデータは業界標準のFASTQ形式で出力されるため、既存のシーケンスプラットフォーム用に構築されたデータ解析パイプラインは、多くの場合そのまま動作します。
提供アプリケーション
(1)全ゲノムシーケンス(WGS)
(2)RNAシーケンス(stranded mRNA-seq)
(3)stLFR (single tube Long Fragment Read) シーケンス
分子バーコード技術により、同じ一分子DNAから得られた複数のライブラリー断片に共通のバーコード配列を付加します。
これにより高精度なショートリード配列でlong rangeの解析を行うことが可能になるため、de novoアセンブリーだけでなく、フェージングや構造変異解析にも有効なデータを得ることができます。
(4)ライブラリー提出によるレーンシーケンス
お客様が調製したライブラリーを提出いただき、シーケンシングのみを提供いたします。
10X ChromiumによるシングルセルRNA-seqライブラリー(scRNA-seq)もシーケンス可能です。
解析の流れ
gDNAまたはRNAサンプルをご送付いただきます。
ご送付いただいたサンプルのクオリティを確認いたします。
各種アプリケーション用のライブラリーを調製いたします。
DNBSEQ-G400RS(MGISEQ-2000RS)システム
またはDNBSEQ-T7システムでのシーケンス
オプションで各種データ解析を承ります。

